Prestations
Cytogénétique conventionnelle (CC)
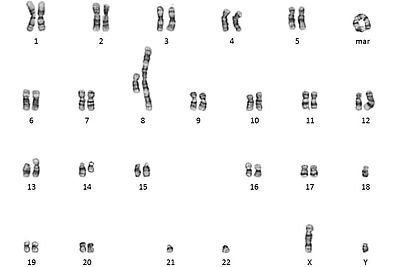
La cytogénétique conventionnelle en bandes G consiste à analyser l’ensemble des chromosomes à une résolution d’environ 10 Mb.
Puce ADN (SNP Array)
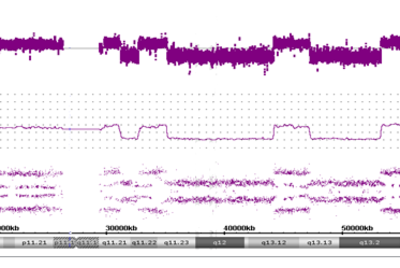
L’analyse par puce ADN permet la détection des anomalies cytogénétiques non-balancées dans l’ensemble du génome et à une résolution élevée.
ddPCR ou PCR digitale
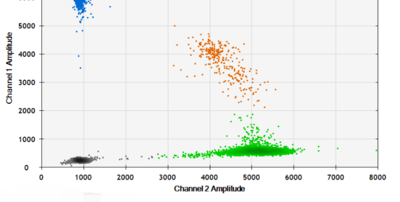
La méthode Digital Droplet PCR est utilisée pour la quantification ultrasensible et absolue des acides nucléiques.
MLPA

L’analyse MLPA est une méthode moléculaire semi-quantitative qui sert à identifier des délétions et des duplications d'un ou plusieurs exons.
Optical Genome Mapping (OGM)
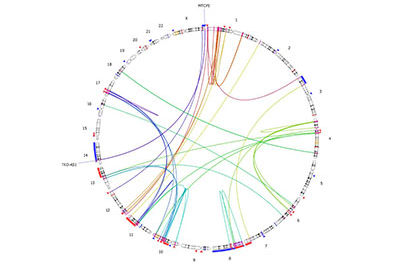
L’analyse par Optical Genome Mapping (OGM) permet la détection d’anomalies de nombre et de structure dans le génome entier (détection de réarrangements balancés et non balancés, détection de variation dans le nombre de copies).
Hybridation in situ fluorescente (FISH)

L’hybridation in situ fluorescente permet l’analyse de régions précises au moyen de sondes spécifiques dans les chromosomes ou dans les noyaux.
Analyse par séquençage à haut débit (NGS)
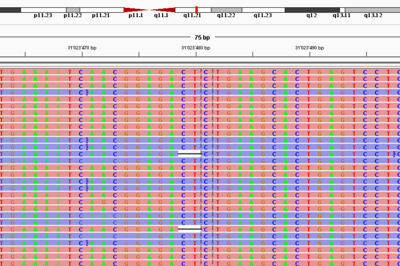
Le séquençage à haut débit permet le séquençage rapide de milliers à des millions de molécules d'ADN ou d’ARN simultanément, en déterminant l'ordre unique et spécifique des bases des acides nucléiques.
Analyse de fragments
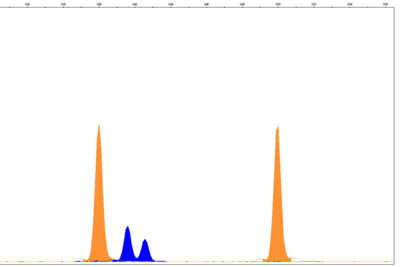
L'analyse de fragments est une méthode moléculaire dans laquelle des fragments fluorescents d'ADN sont séparés par électrophorèse capillaire et comparés à un marqueur afin de déterminer leur taille.
Sanger
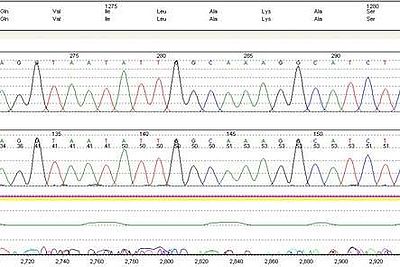
Le séquençage Sanger est une méthode moléculaire qui détermine l’ordre unique et spécifique des bases d’ADN en comparant la séquence du patient à une séquence de référence.




